データの準備#
データの準備
h5 形式を cbf 形式に変換します:XDS は h5 形式の画像を処理できないため、プロセスの前に画像を cbf 形式に変換する必要があります。macOS では、eiger2cbfというプログラムが便利です。
eiger2cbf path/to/your/h5_master_data -- フレーム数を取得
eiger2cbf path/to/your/h5_master_data N out.cbf -- N番目のフレームをout.cbfに書き込む
eiger2cbf path/to/your/h5_master_data N -- N番目のフレームをSTDOUTに書き込む
eiger2cbf path/to/your/h5_master_data N:M out -- NからM番目のフレームをoutNNNNNN.cbfに書き込む
例:
eiger2cbf path/to/your/h5_master_data 1:720 out # すべての720枚の画像をcbf形式に変換する
X 線データの処理#
XDS で処理する#
XDSGUI
-
この手順に従って XDSGUI をインストールします。
-
ターミナルで XDSGUI を実行します。
cd path/to/your/images xdsgui -
画像をロードし、XDS.INP ファイルを生成します:
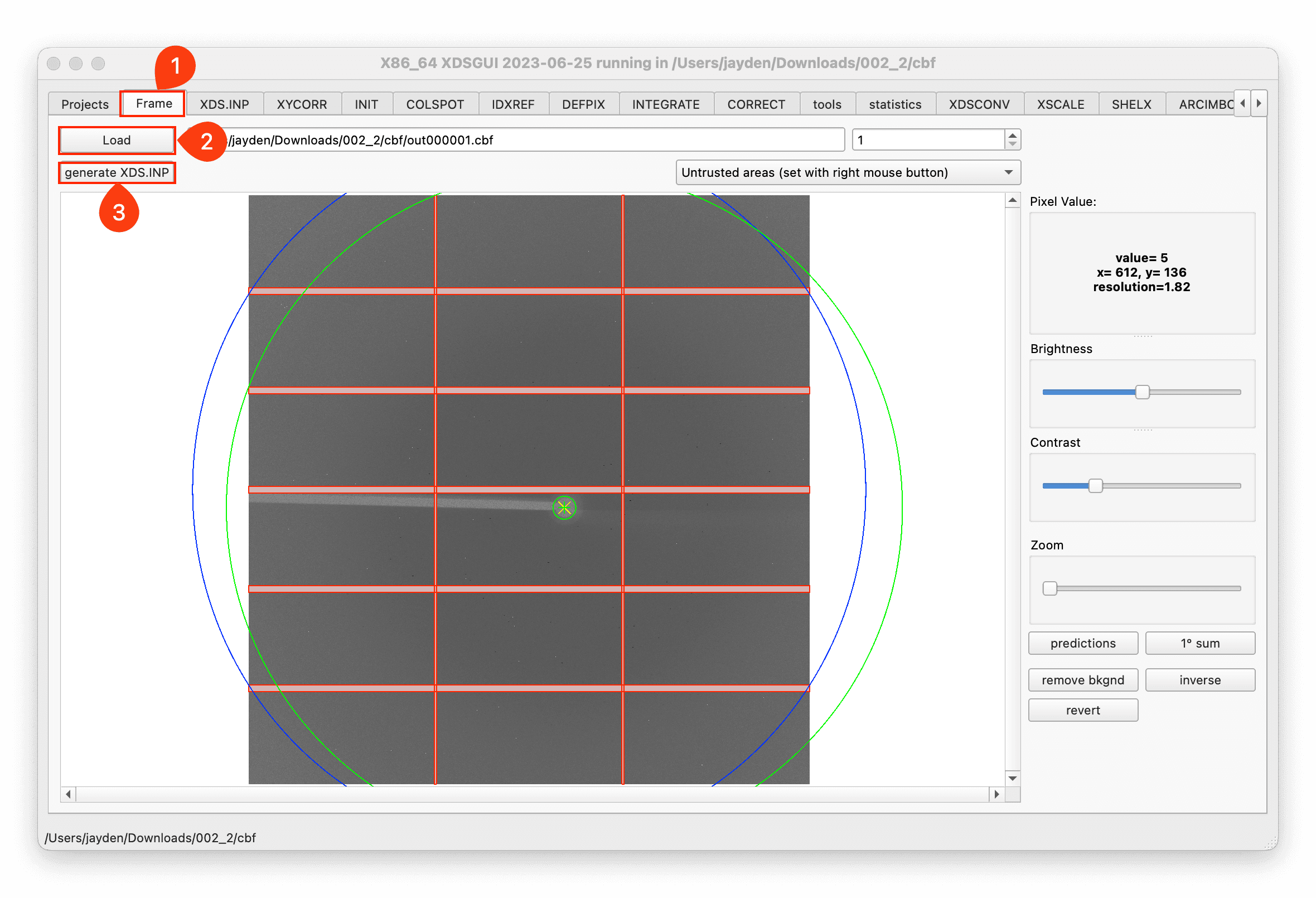
-
XDS.INP ファイルを必要に応じて編集し、
Run XDSをクリックします。SSRF の 10U2 ビームラインでデータを収集した場合、
ROTATION_AXISパラメータを0 -1 0に変更する必要があります。 -
CORRECT ページで結果を確認します。
Xia2 で処理する#
-
Xia2 を使用する方法は 2 つあります:CCP4 で Xia2 を使用するか、最新の xia2/DIALS バンドルを **このウェブサイト** に従ってインストールします。
-
以下のコマンドでデータを処理します:
cd path/to/images xia2 pipeline=dials-aimless ./ goniometer.axes=0.000000,1.000000,0.000000 xia2.settings.resolution.d_min=2
autoPROC で処理する#
-
ライセンスを申請し、autoPROC を **このウェブサイト** に従ってインストールします。
-
以下のコマンドでデータを処理します:
cd path/to/images process -I . -d autoproc -R 50 2 > out.log # 結果をautoprocという名前のサブディレクトリに保存し、解像度は2から50まで変化し、ログをout.logファイルに保存します。
構造の決定#
データの検証#
-
autoproc ディレクトリ内の
XDS_ASCII.HKLファイルを回折ファイルとして見つけます。 -
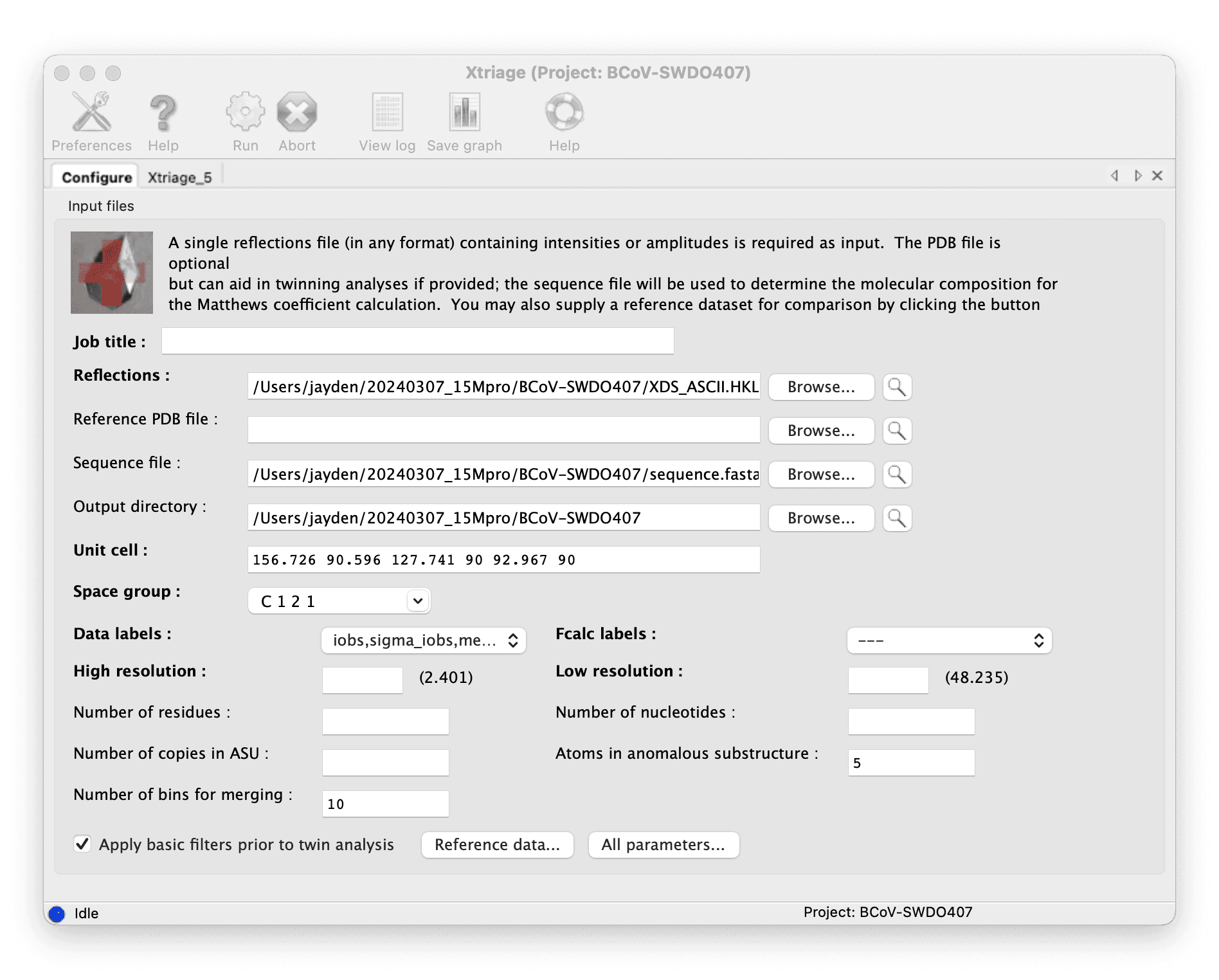
Phenix Xtriage でデータを検証し、回折ファイルを入力して実行します。
分子置換#
-
autoproc ディレクトリ内の
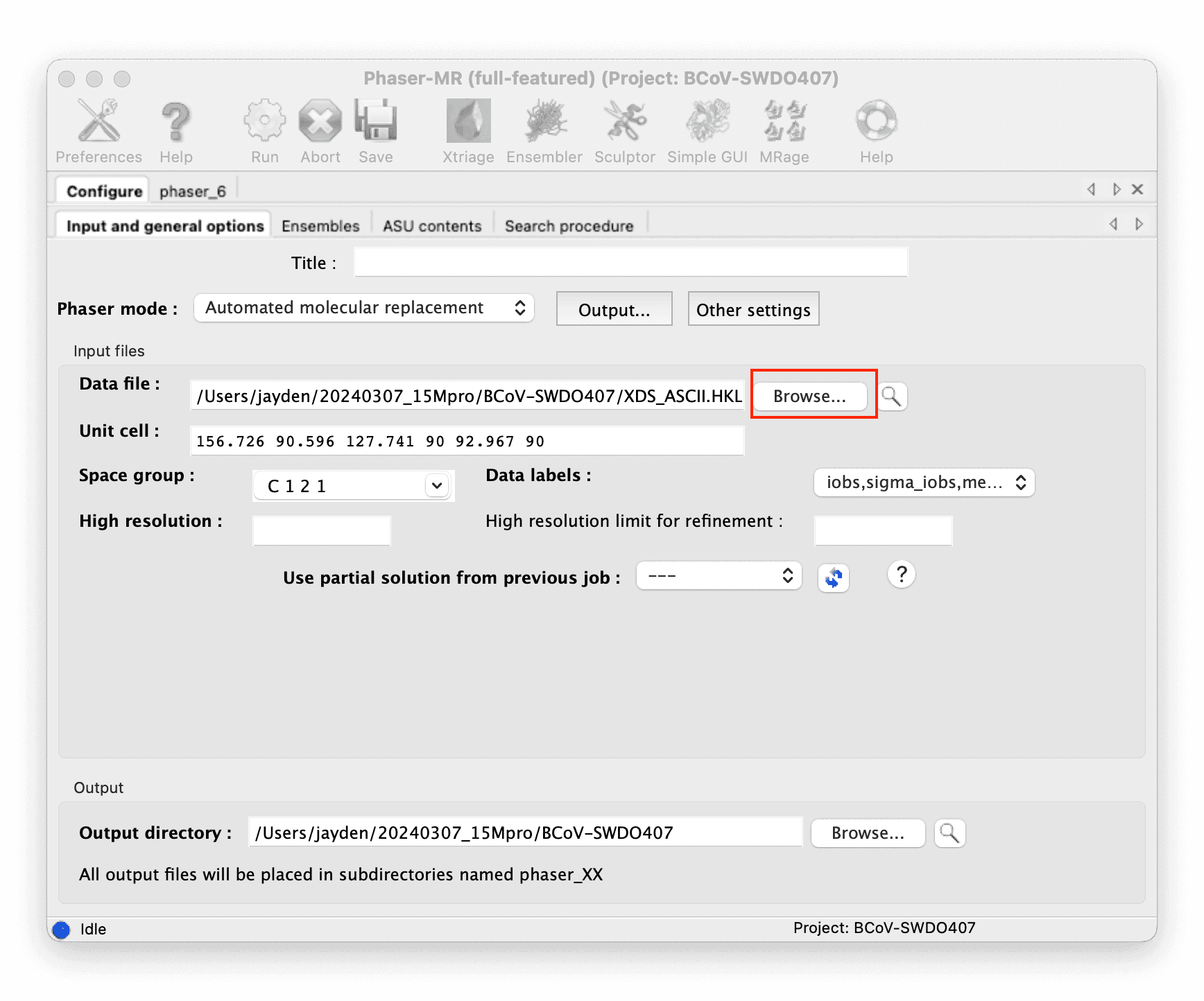
XDS_ASCII.HKLファイルを回折ファイルとして見つけます。 -
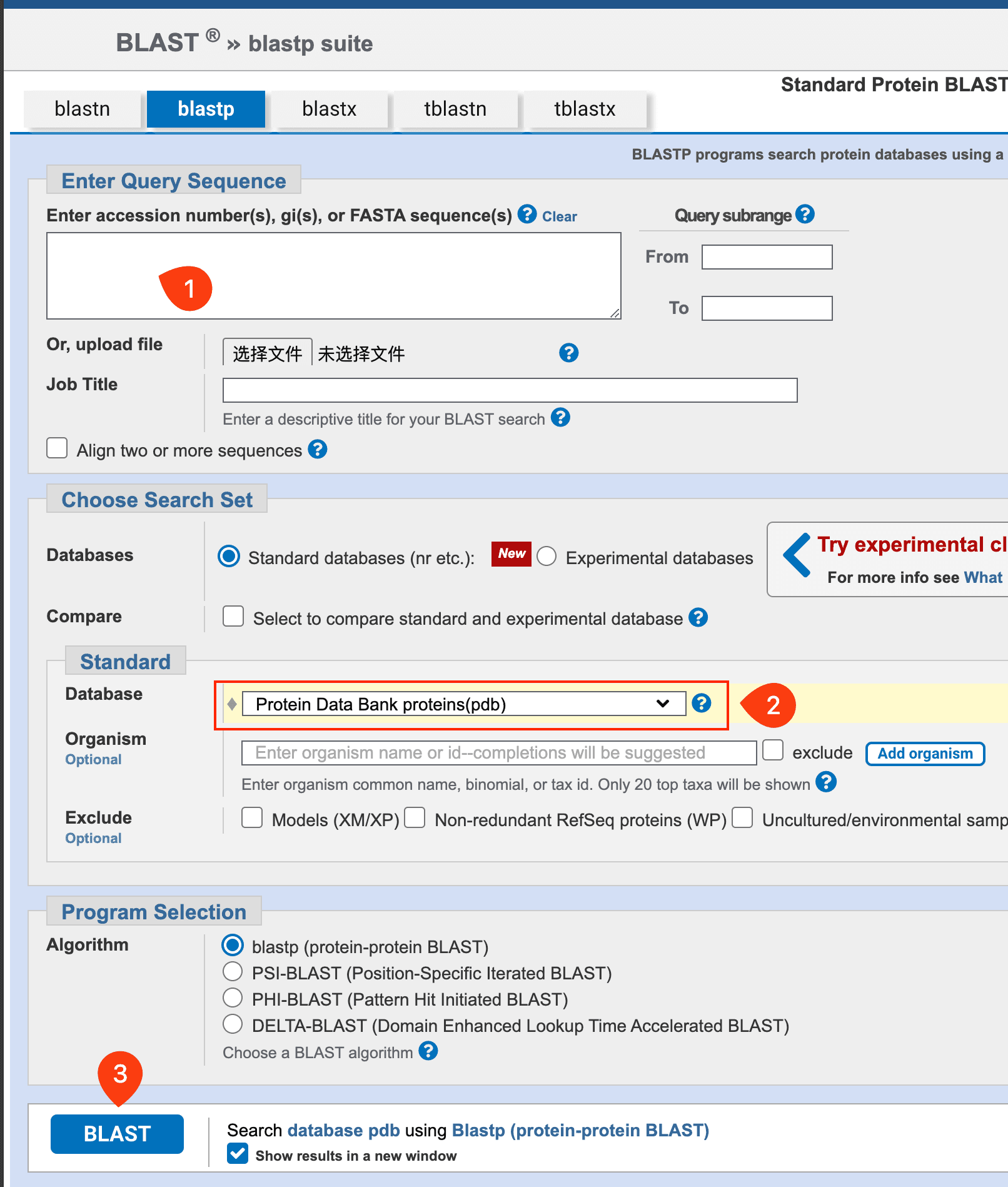
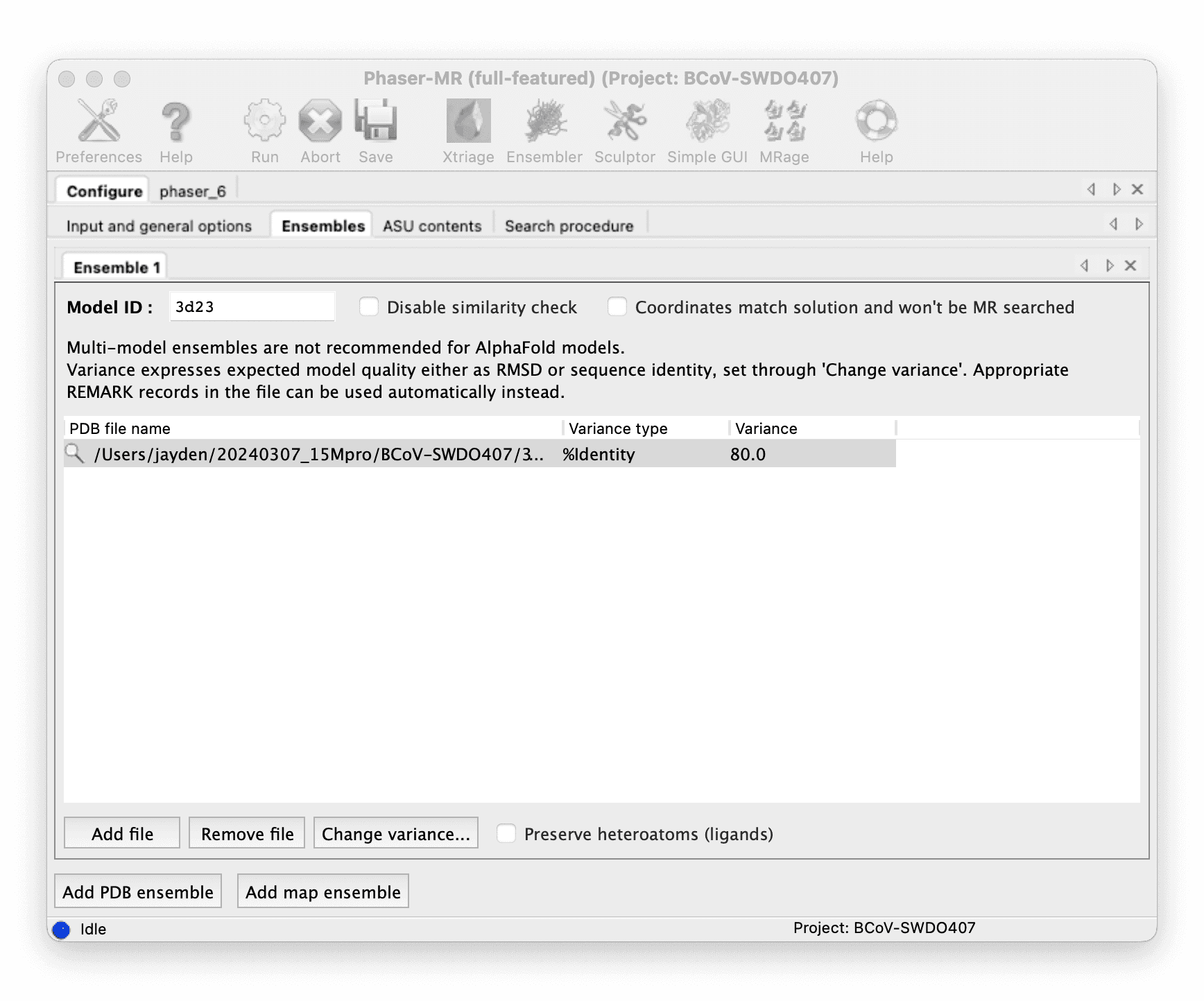
テンプレートモデルを取得します:
- タンパク質の配列を PDB データベースで検索し、適切な構造を選択して PDB ファイルをダウンロードします。
- テンプレートモデルをダウンロードした後、冗長なコピーと水分子を削除するために PyMOL で編集する必要があります。
-
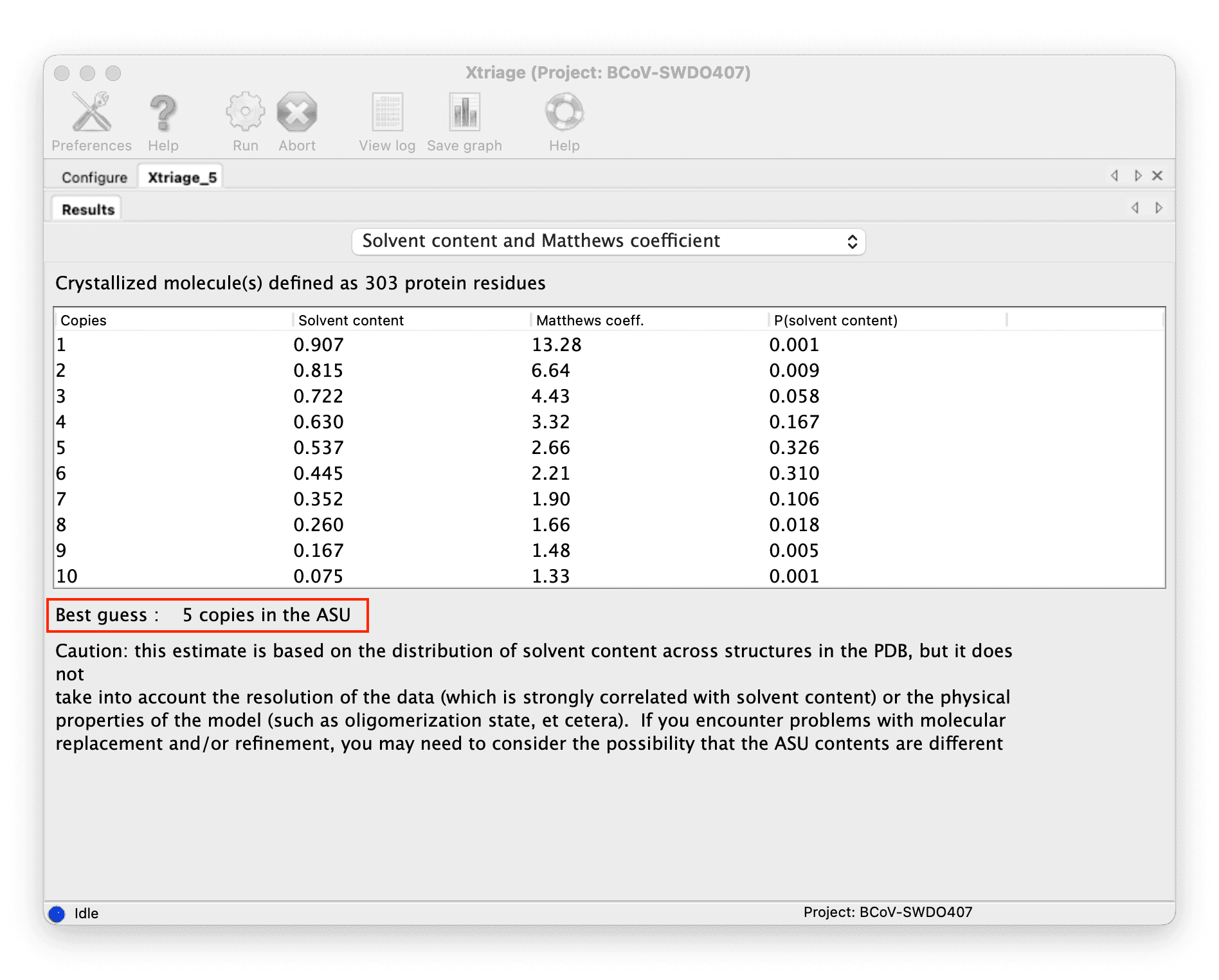
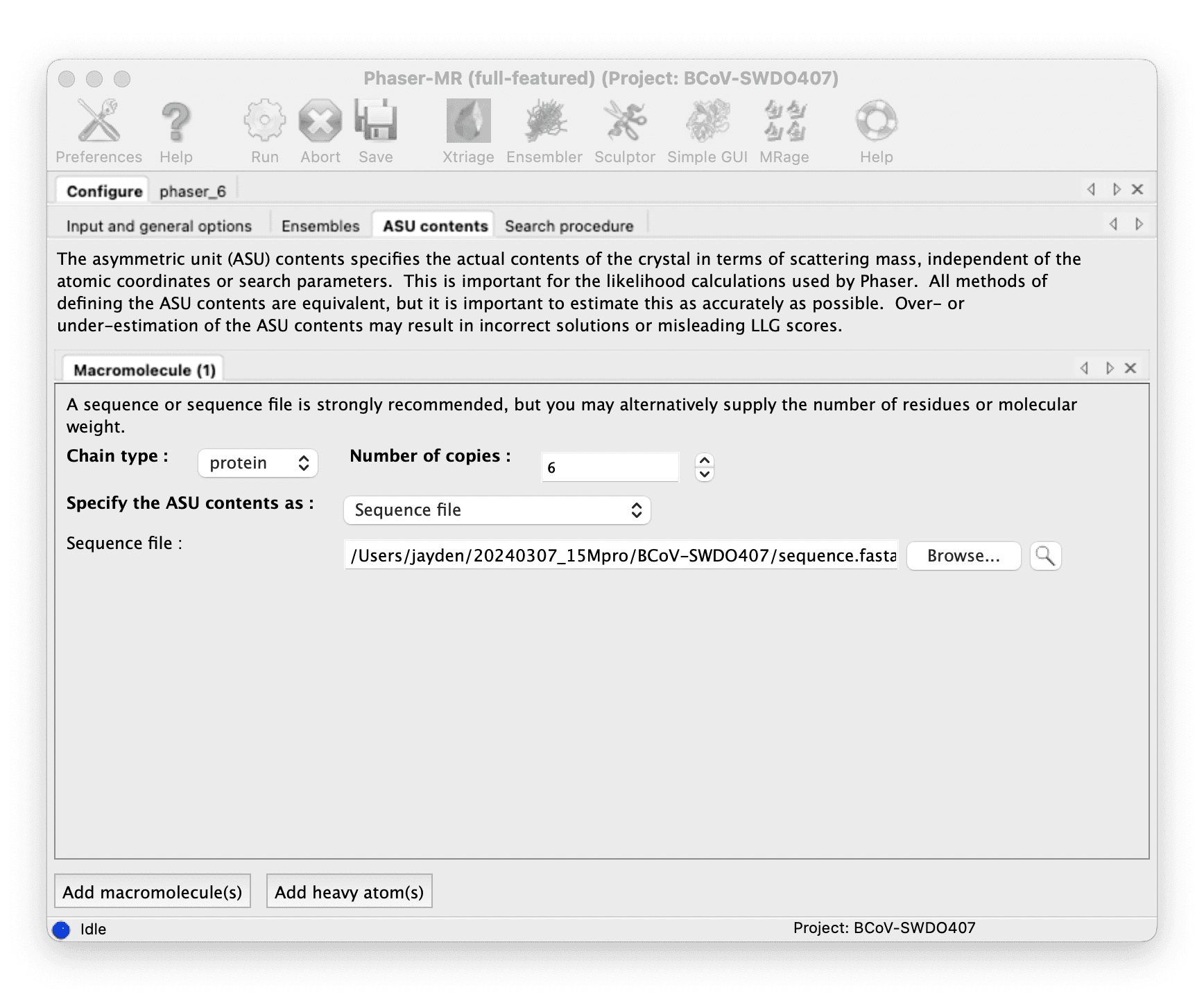
ASU(非対称単位)にいくつのコピーがあるかを決定します:
-
Xtriage の結果から数を取得できます。
::: grid {cols=2,rows=1,gap=12,type=images}
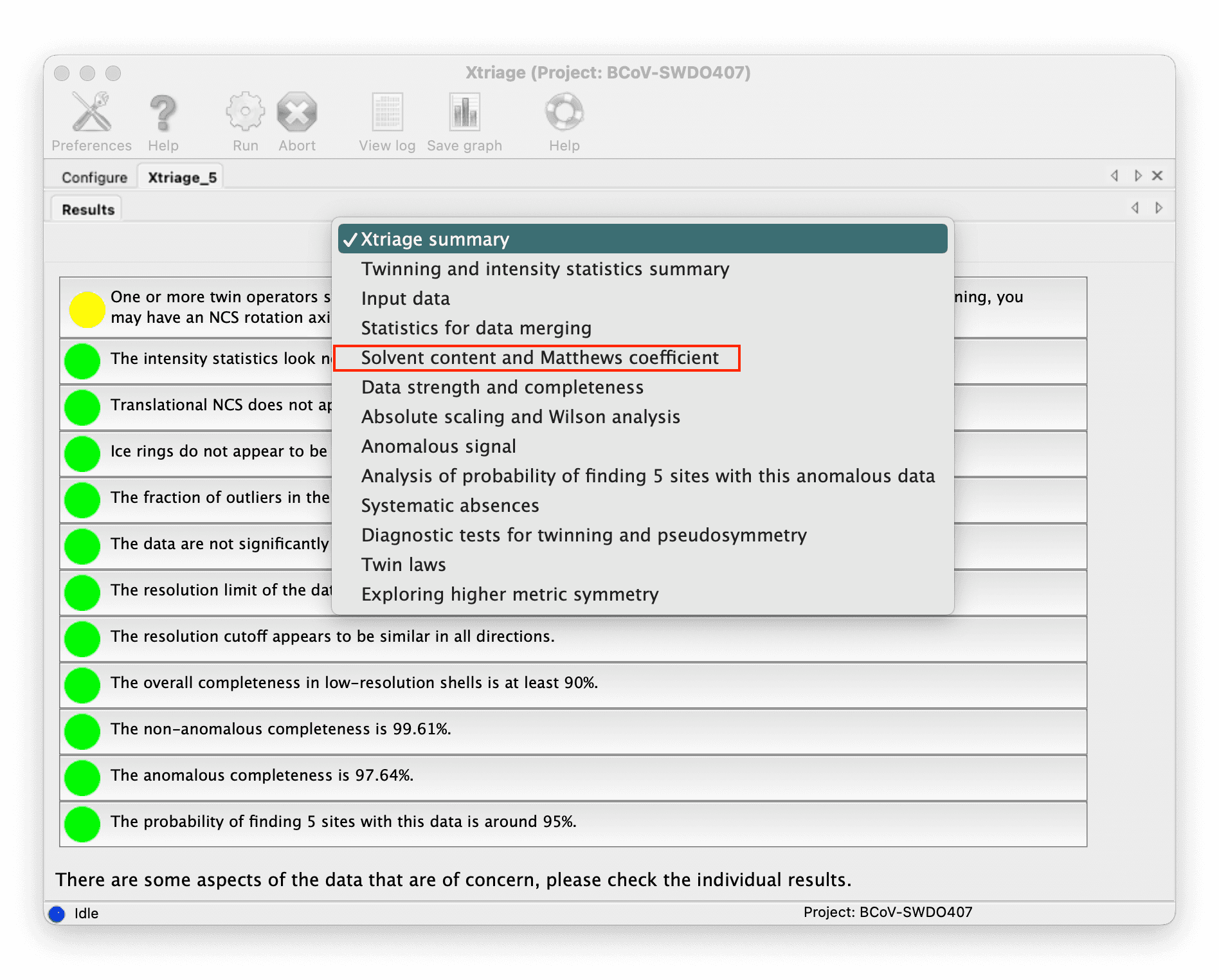
::: -
または、CCP4 のセル内容解析ツールを使用することもできます。
[!NOTE]
溶媒含有量を通じてコピーの数を決定できます。一般的に、ほとんどの生物マクロ分子結晶の溶媒含有量は 40%から 60%の間です。
-
-
Phenix Phaser-MR を使用して分子置換を行います。
::: grid {cols=4,rows=1,gap=12,type=images}
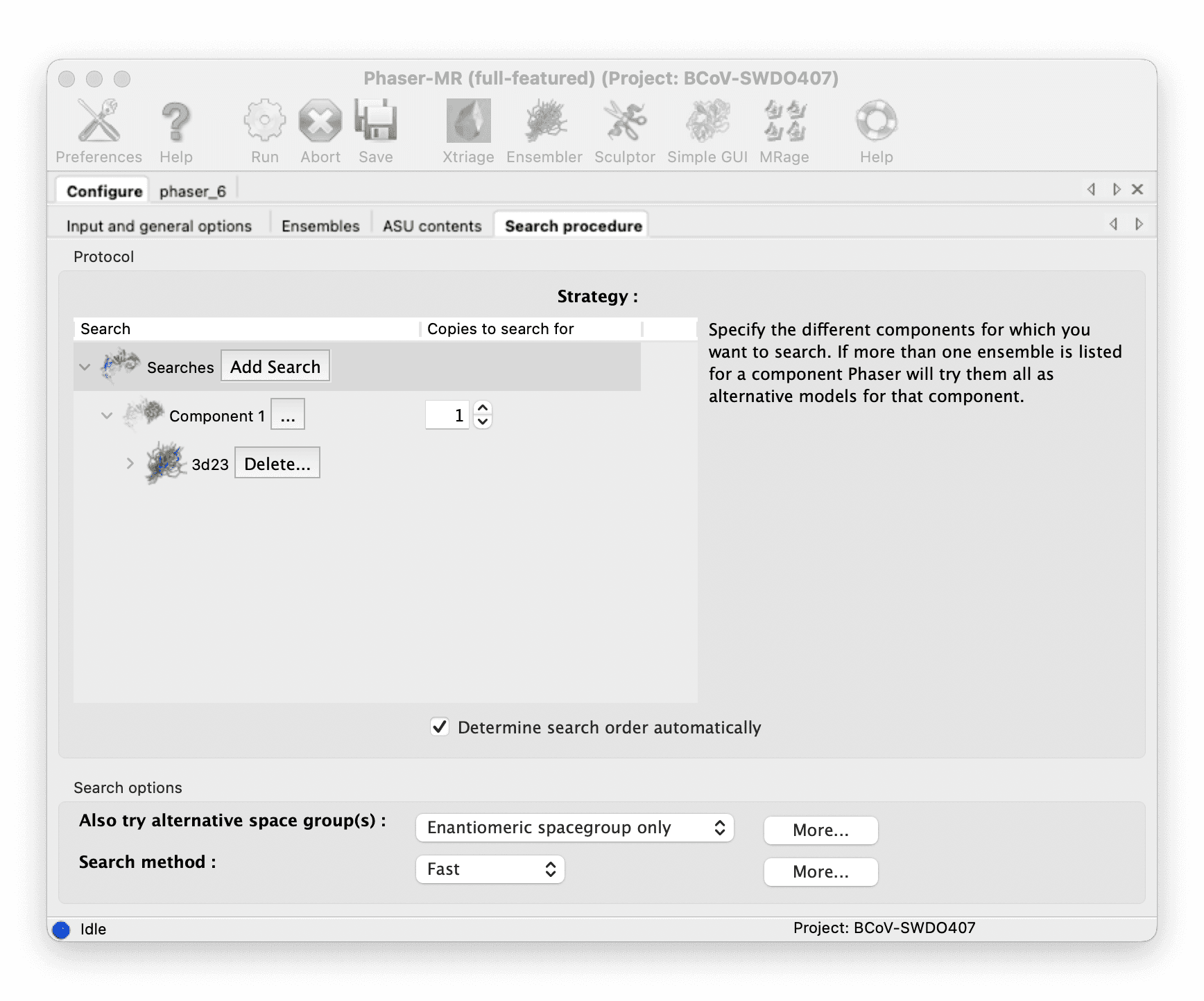
::: -
Phenix AutoBuild を使用してモデルを構築します:分子置換の結果を入力し、実行します。
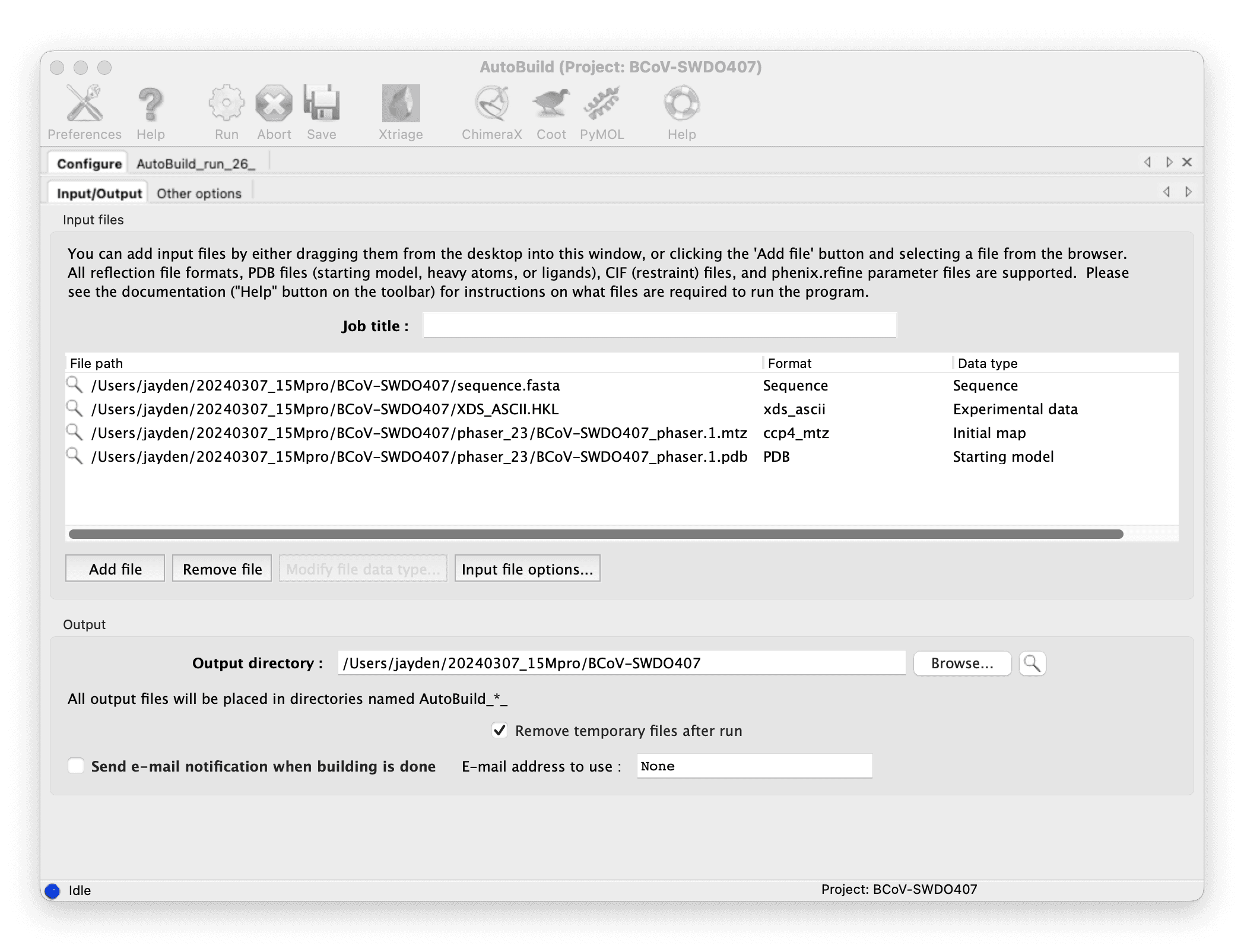
-
Coot で AutoBuild のモデルを確認し、手動で精製します。
::: grid {cols=2,rows=1,gap=12,type=images}
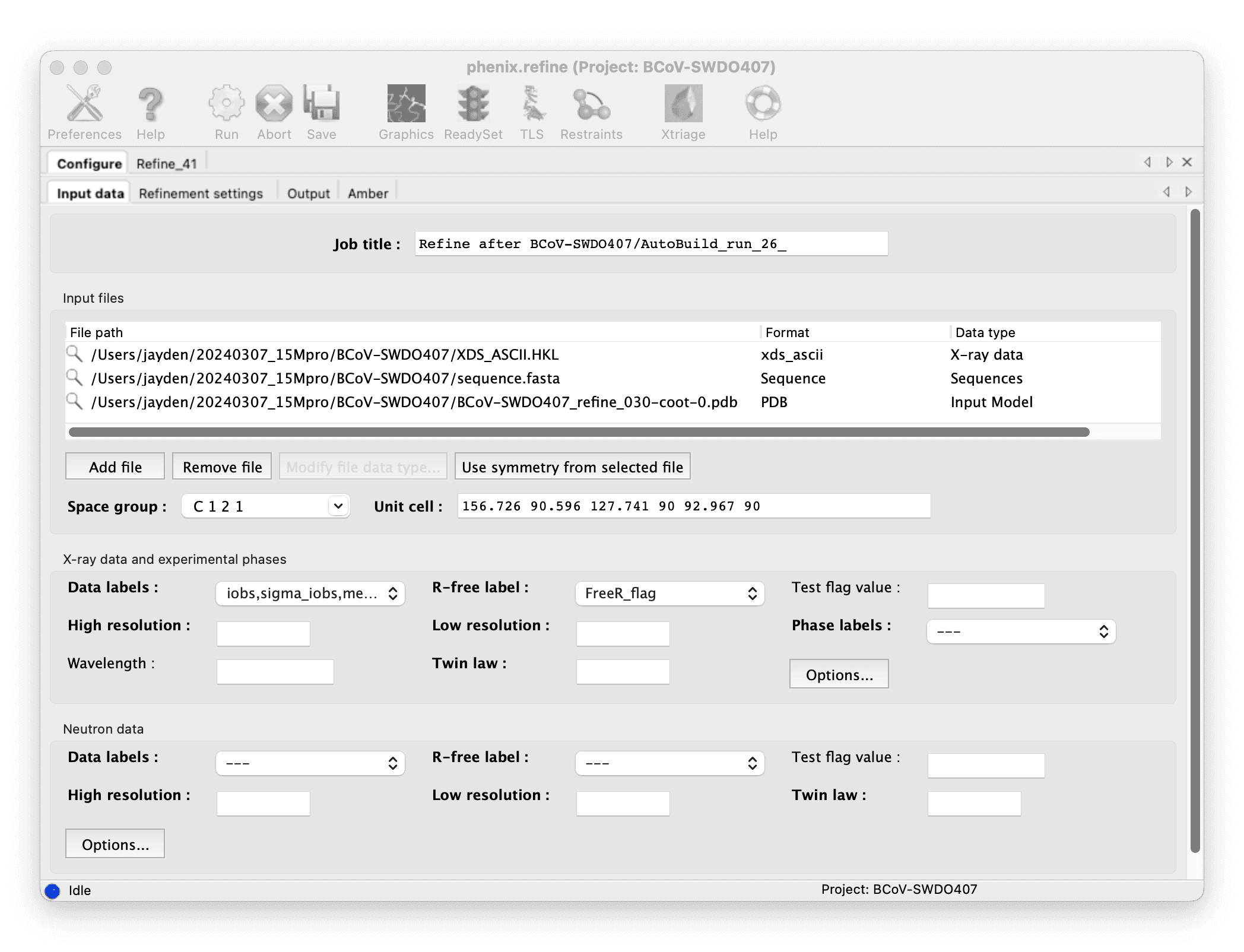
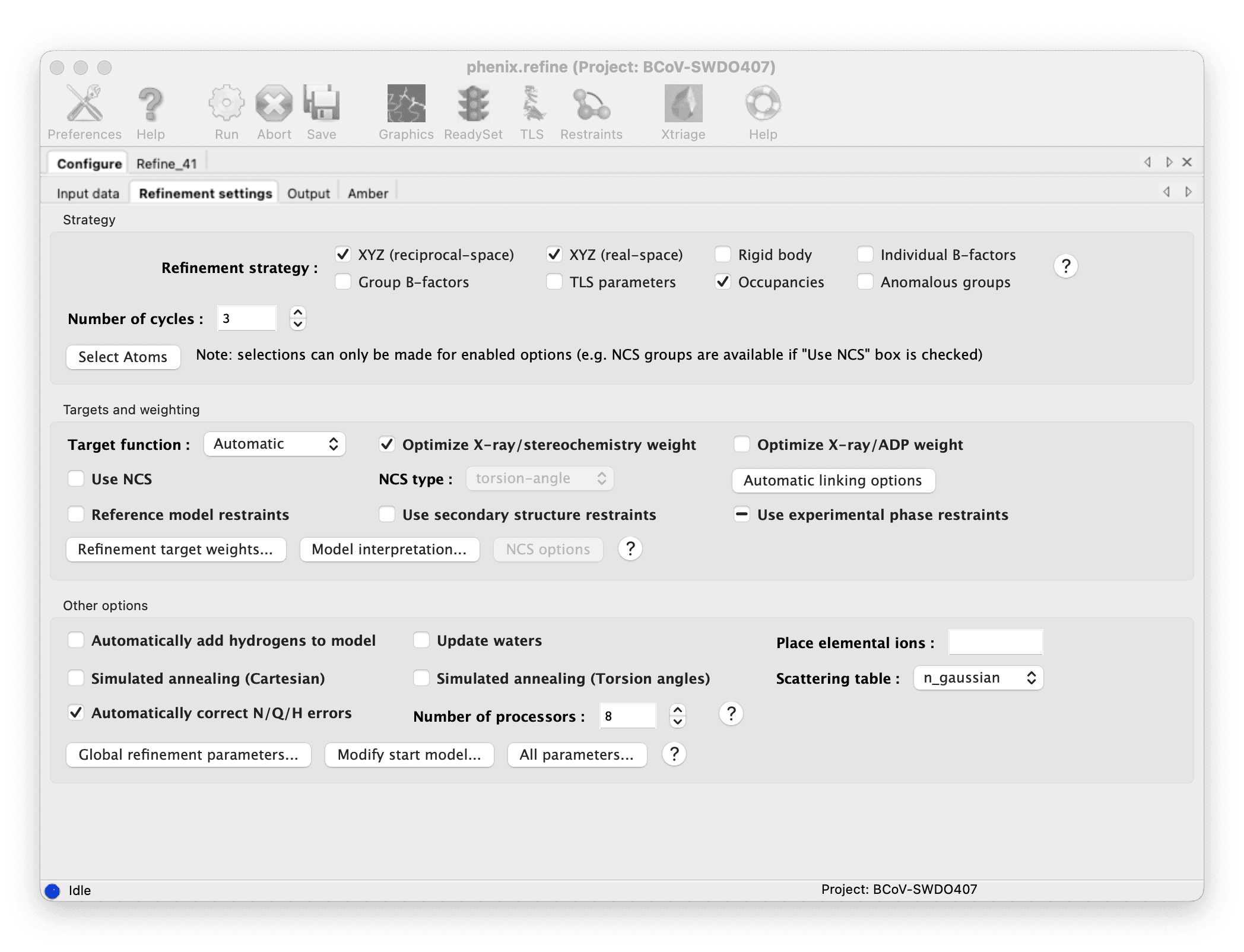
:::
この文書は Mix Space からの同期更新であり、xLog にも掲載されています。
オリジナルのリンクは https://xxu.do/posts/academic/X-ray-data-processing です。