准备数据#
将 h5 格式转换为 cbf 格式:XDS 无法处理 h5 格式的图像,因此您需要在处理之前将图像转换为 cbf 格式。在 macOS 上,eiger2cbf 是一个有用的程序。
eiger2cbf path/to/your/h5_master_data -- 获取帧数
eiger2cbf path/to/your/h5_master_data N out.cbf -- 将第 N 帧写入 out.cbf
eiger2cbf path/to/your/h5_master_data N -- 将第 N 帧写入 STDOUT
eiger2cbf path/to/your/h5_master_data N:M out -- 将第 N 帧到第 M 帧写入 outNNNNNN.cbf
例如:
eiger2cbf path/to/your/h5_master_data 1:720 out # 将所有 720 张图像转换为 cbf 格式
X 射线数据处理#
使用 XDS 处理#
XDSGUI
-
根据此 说明 安装 XDSGUI。
-
在终端中运行 XDSGUI。
cd path/to/your/images xdsgui -
加载图像并生成 XDS.INP 文件:
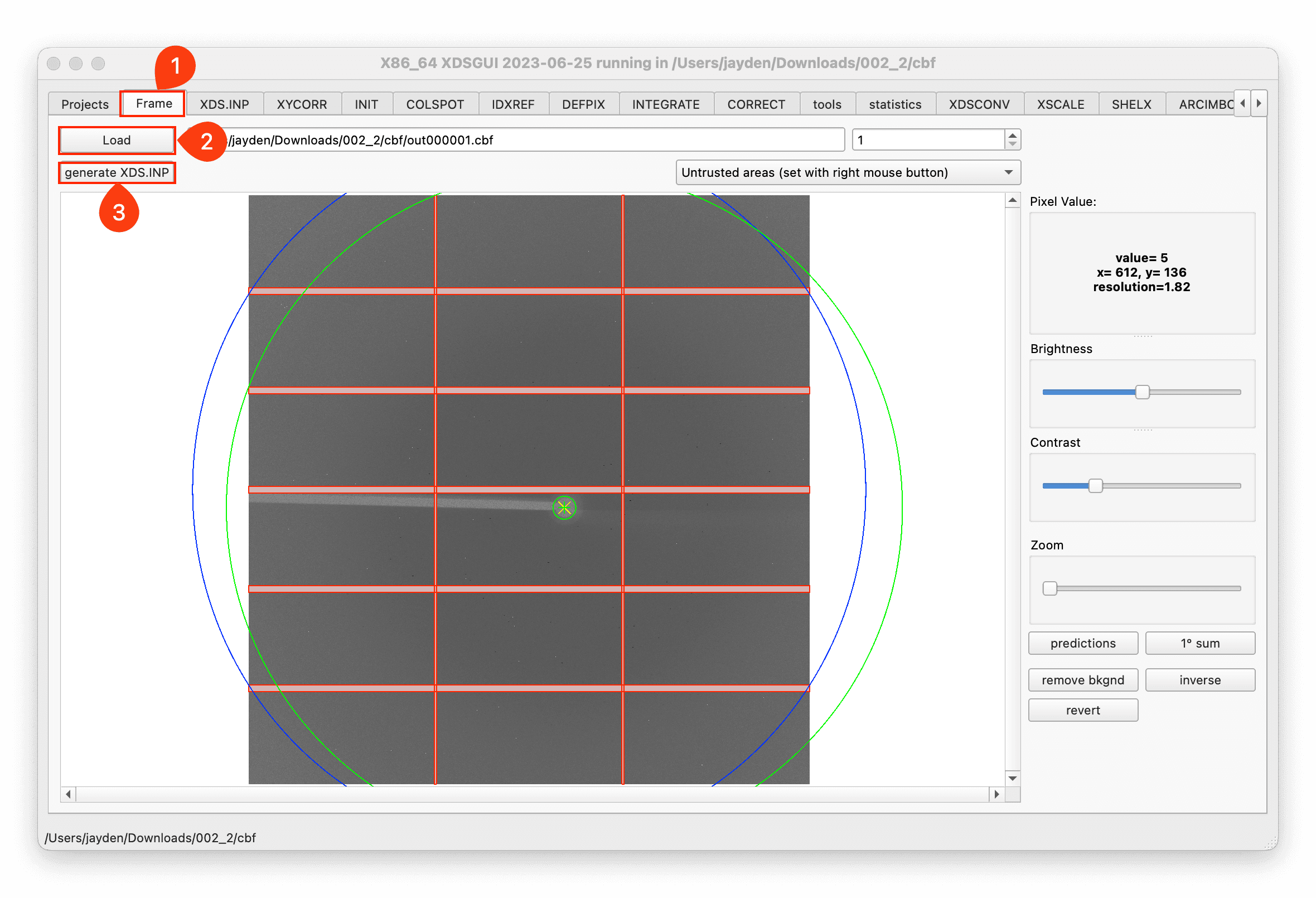
-
修改 XDS.INP 文件以满足您的需求,然后点击
Run XDS。如果您在 SSRF 的 10U2 光束线收集数据,您应该将
ROTATION_AXIS参数修改为0 -1 0。 -
在 CORRECT 页面检查结果。
使用 Xia2 处理#
-
有两种方法使用 Xia2:在 CCP4 中使用 Xia2,或根据 此网站 安装最新的 xia2/DIALS 套件。
-
使用它处理您的数据:
cd path/to/images xia2 pipeline=dials-aimless ./ goniometer.axes=0.000000,1.000000,0.000000 xia2.settings.resolution.d_min=2
使用 autoPROC 处理#
-
根据 此网站 请求许可证并安装 autoPROC。
-
使用它处理您的数据:
cd path/to/images process -I . -d autoproc -R 50 2 > out.log #将结果保存在名为 autoproc 的子目录中,分辨率范围从 2 到 50,将日志保存到 out.log 文件中。
结构确定#
验证数据#
-
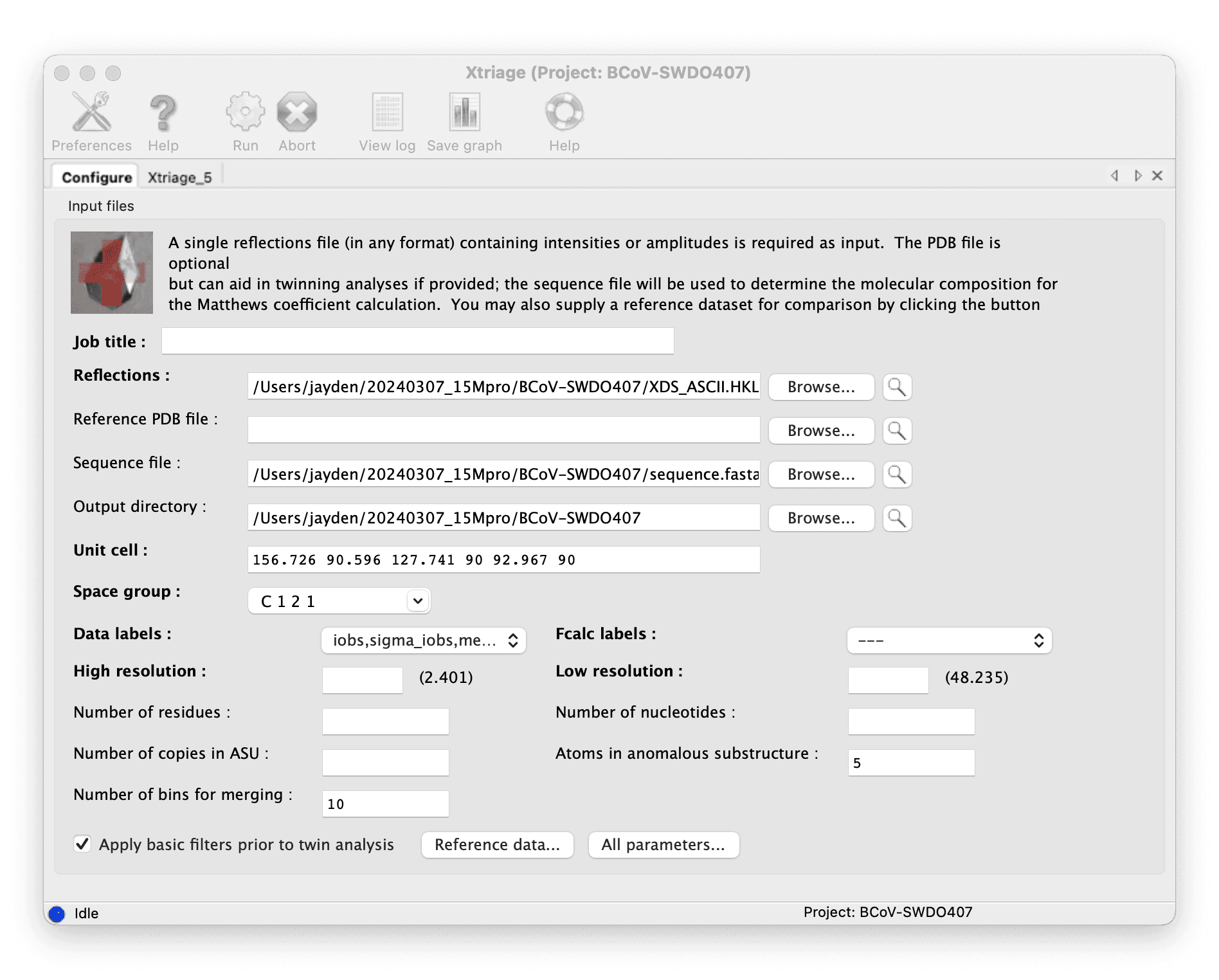
在 autoproc 目录中找到
XDS_ASCII.HKL文件作为衍射文件。 -
使用 Phenix Xtriage 验证数据,将衍射文件放入并运行。
分子替换#
-
在 autoproc 目录中找到
XDS_ASCII.HKL文件作为衍射文件。 -
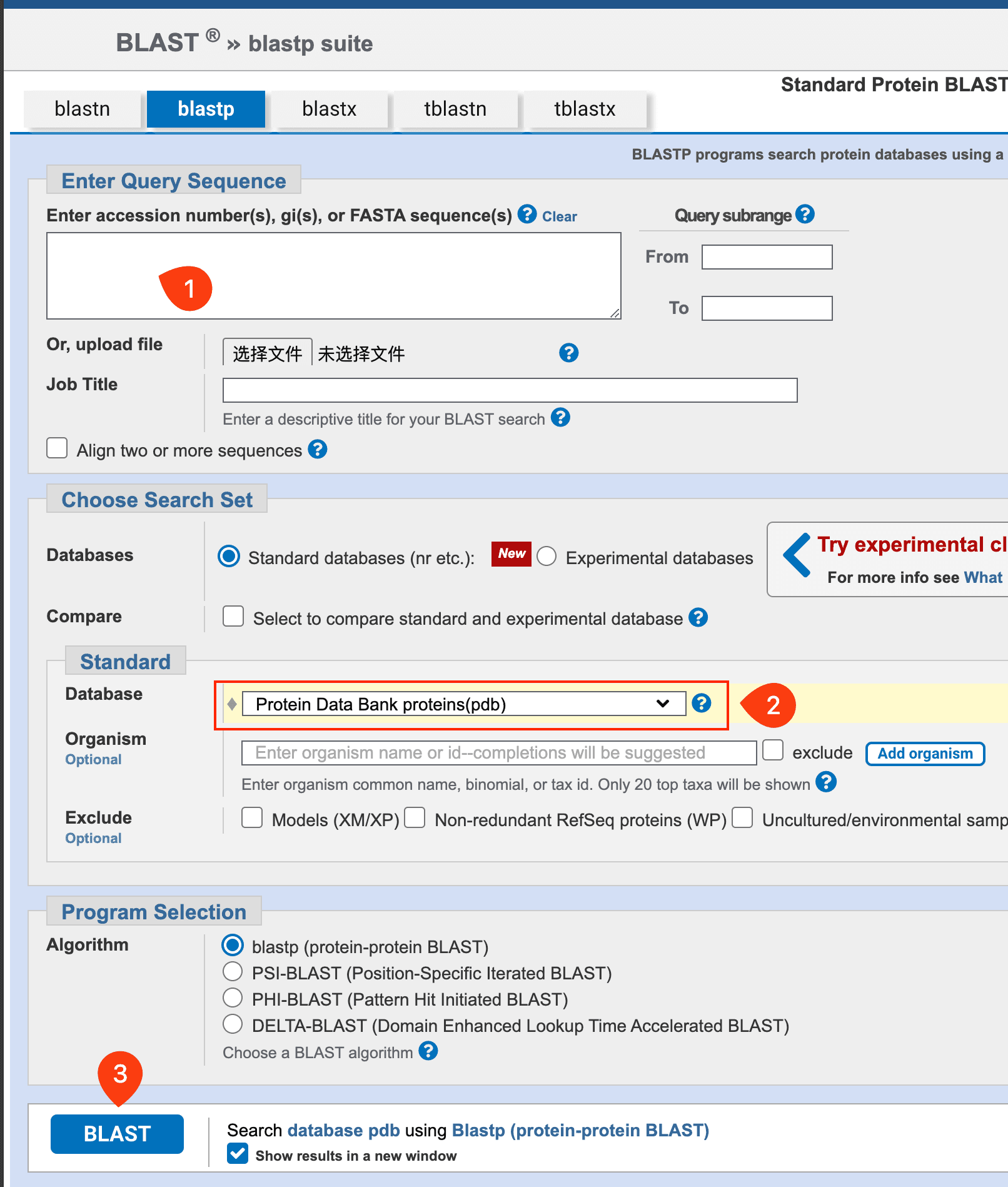
获取模板模型:
- 使用 PDB 数据库对您的蛋白质序列进行 BLAST,选择合适的结构并下载 PDB 文件。
- 下载模板模型后,您需要使用 PyMOL 编辑它以去除冗余副本和水分子。
-
确定 ASU(不对称单元)中有多少个副本:
-
您可以从 Xtriage 的结果中获取数量。
::: grid {cols=2,rows=1,gap=12,type=images}
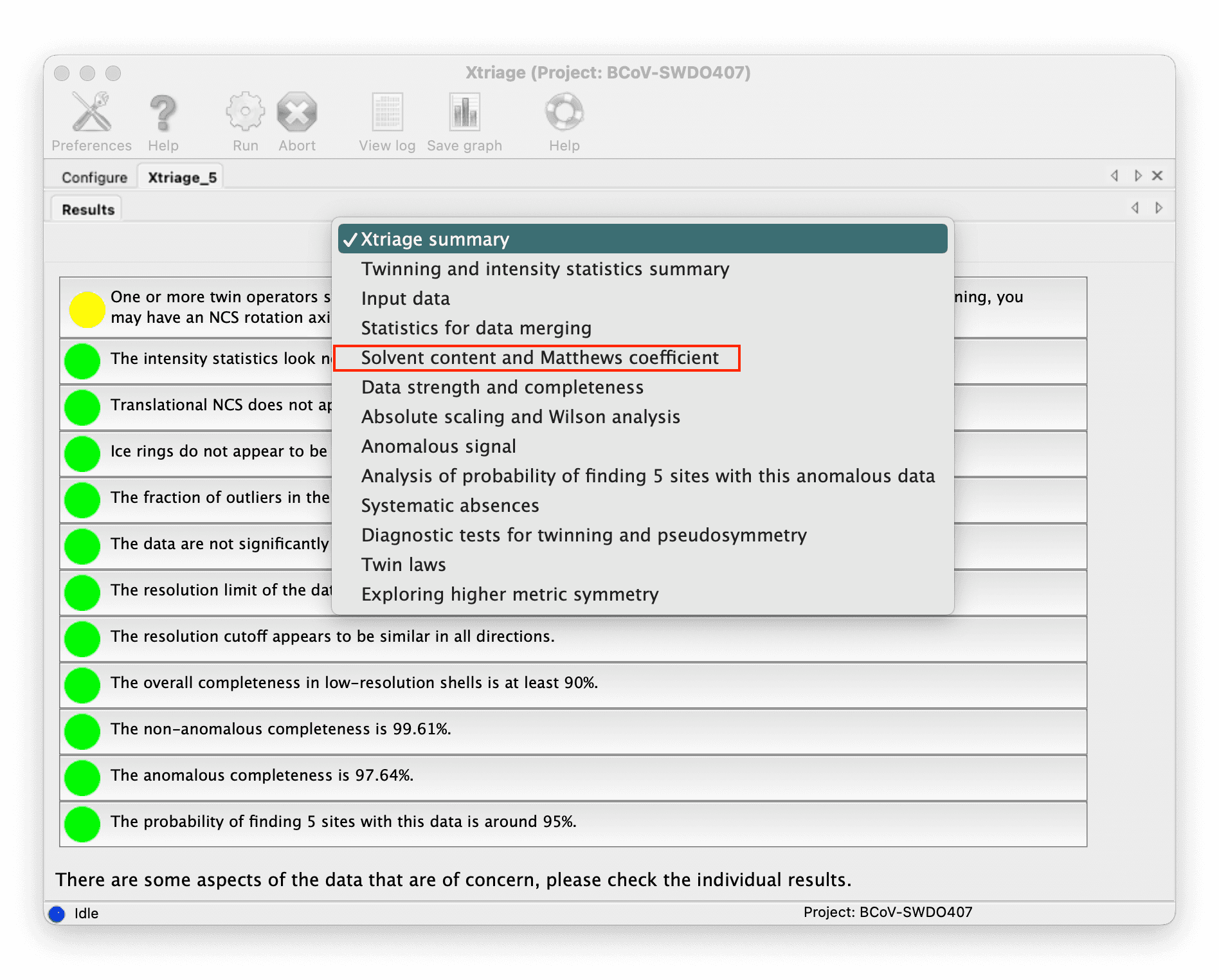
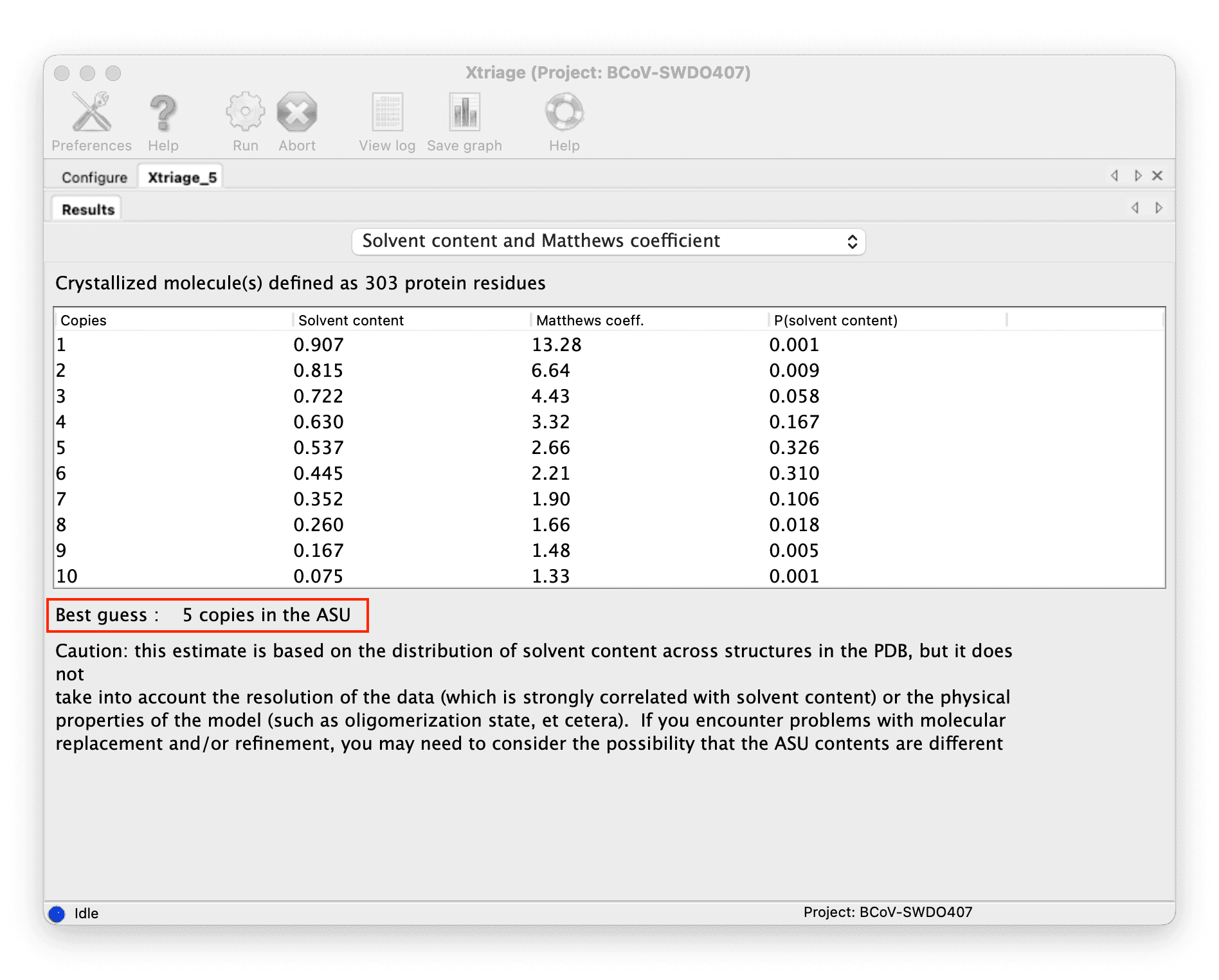
::: -
或者您可以使用 CCP4 中的单元内容分析工具。
[!NOTE]
您可以通过溶剂含量确定副本数量,通常,大多数生物大分子晶体的溶剂含量在 40% 到 60% 之间。
-
-
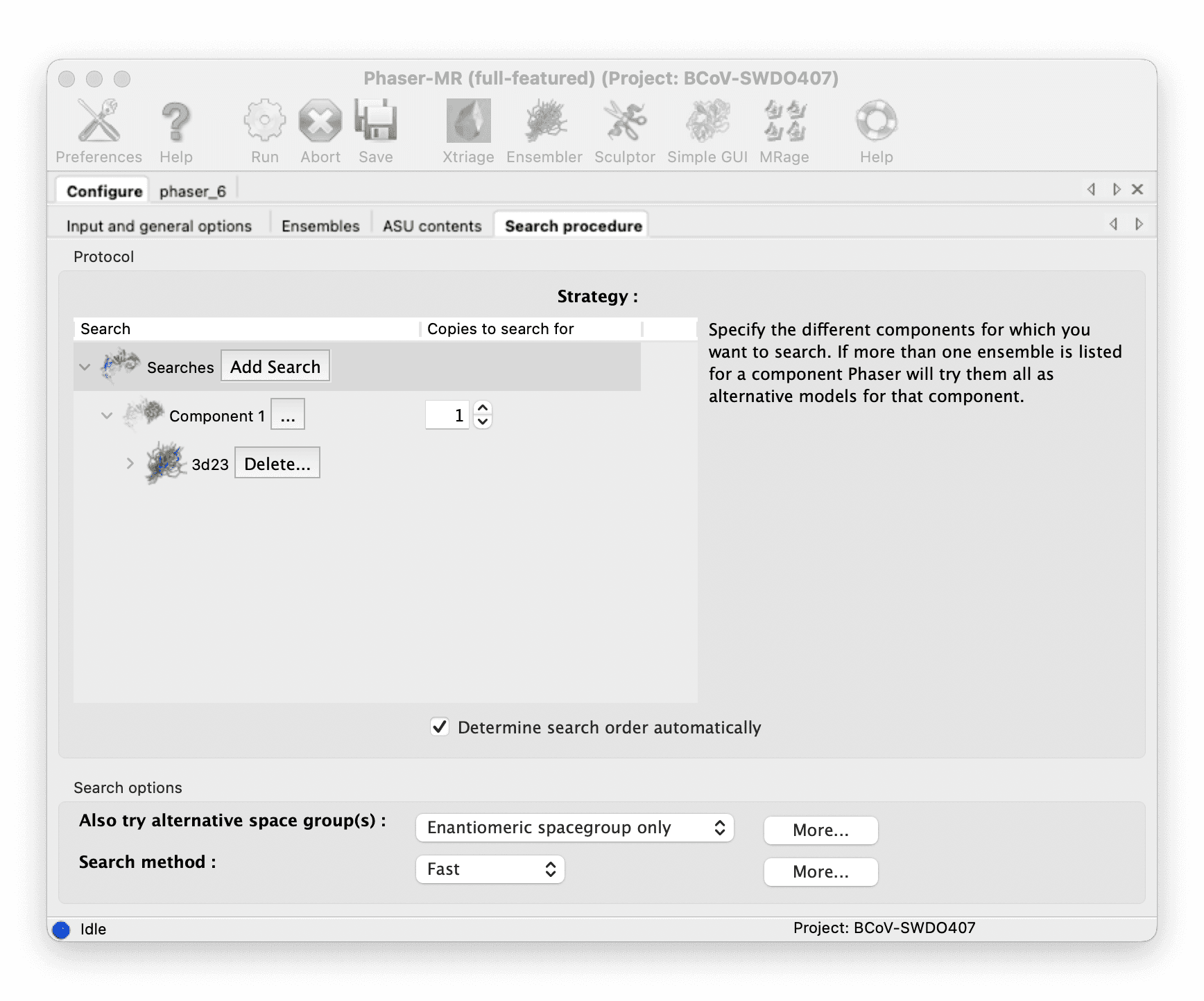
使用 Phenix Phaser-MR 进行分子替换。
::: grid {cols=4,rows=1,gap=12,type=images}
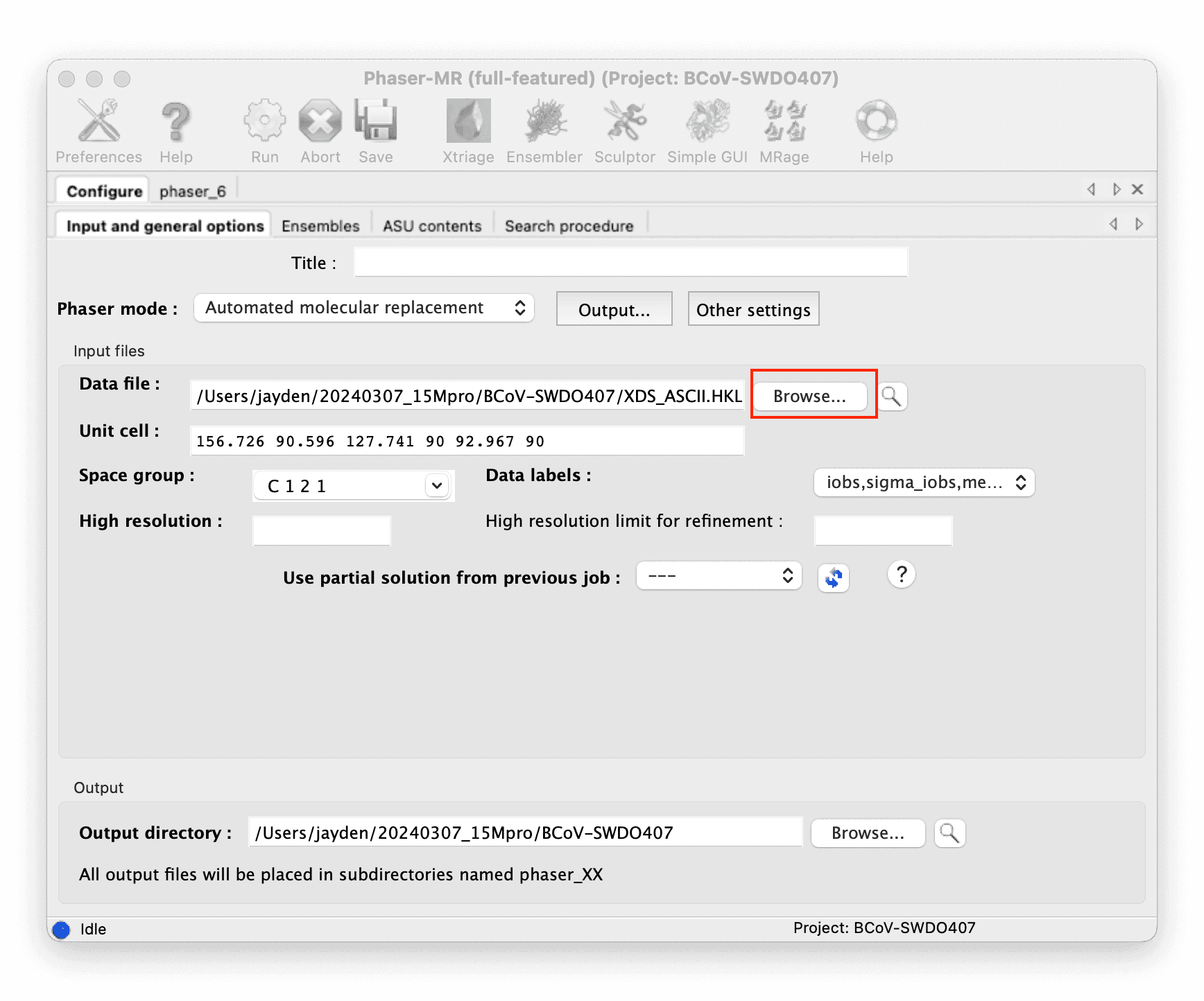
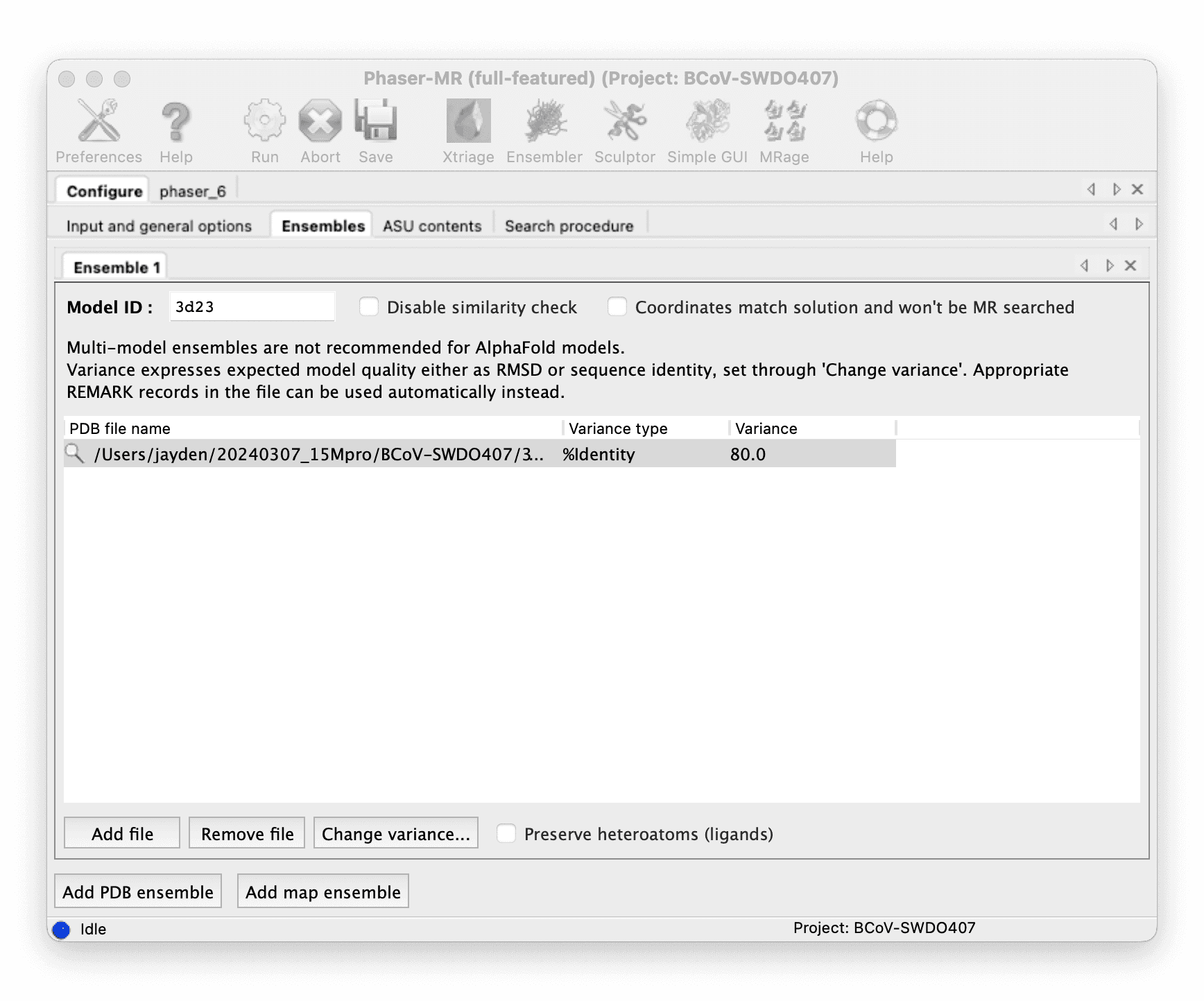
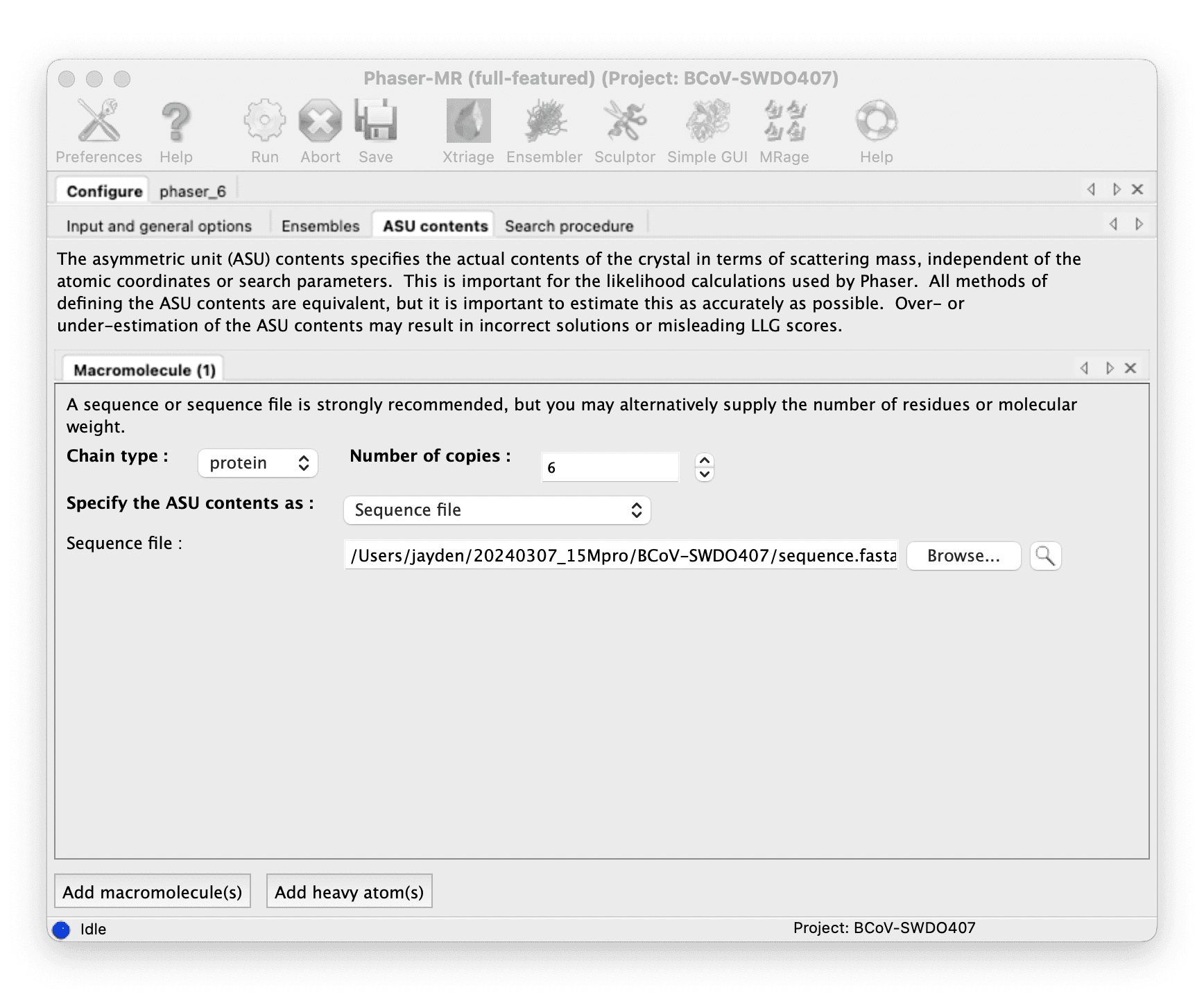
::: -
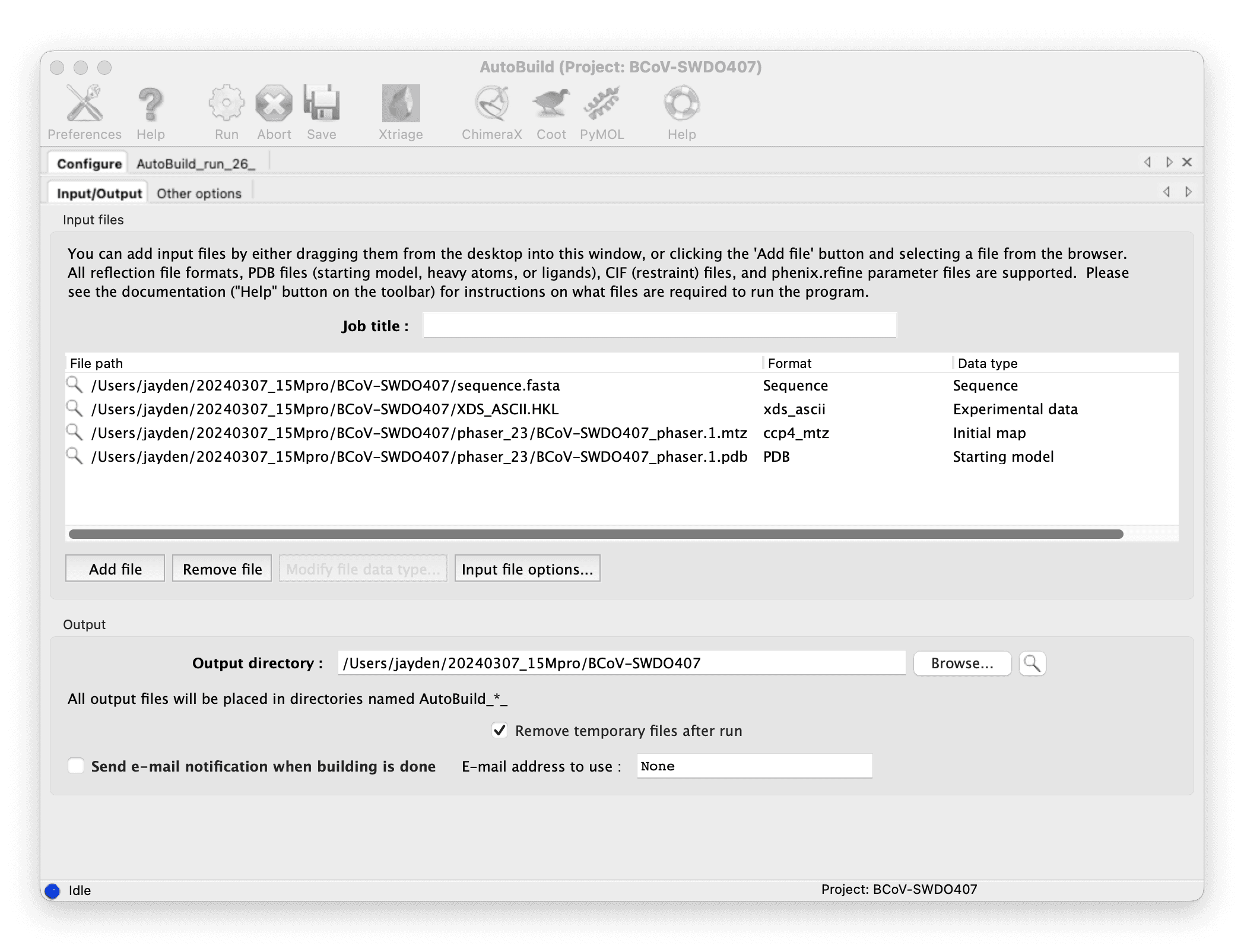
使用 Phenix AutoBuild 构建您的模型:输入分子替换的结果并运行。
-
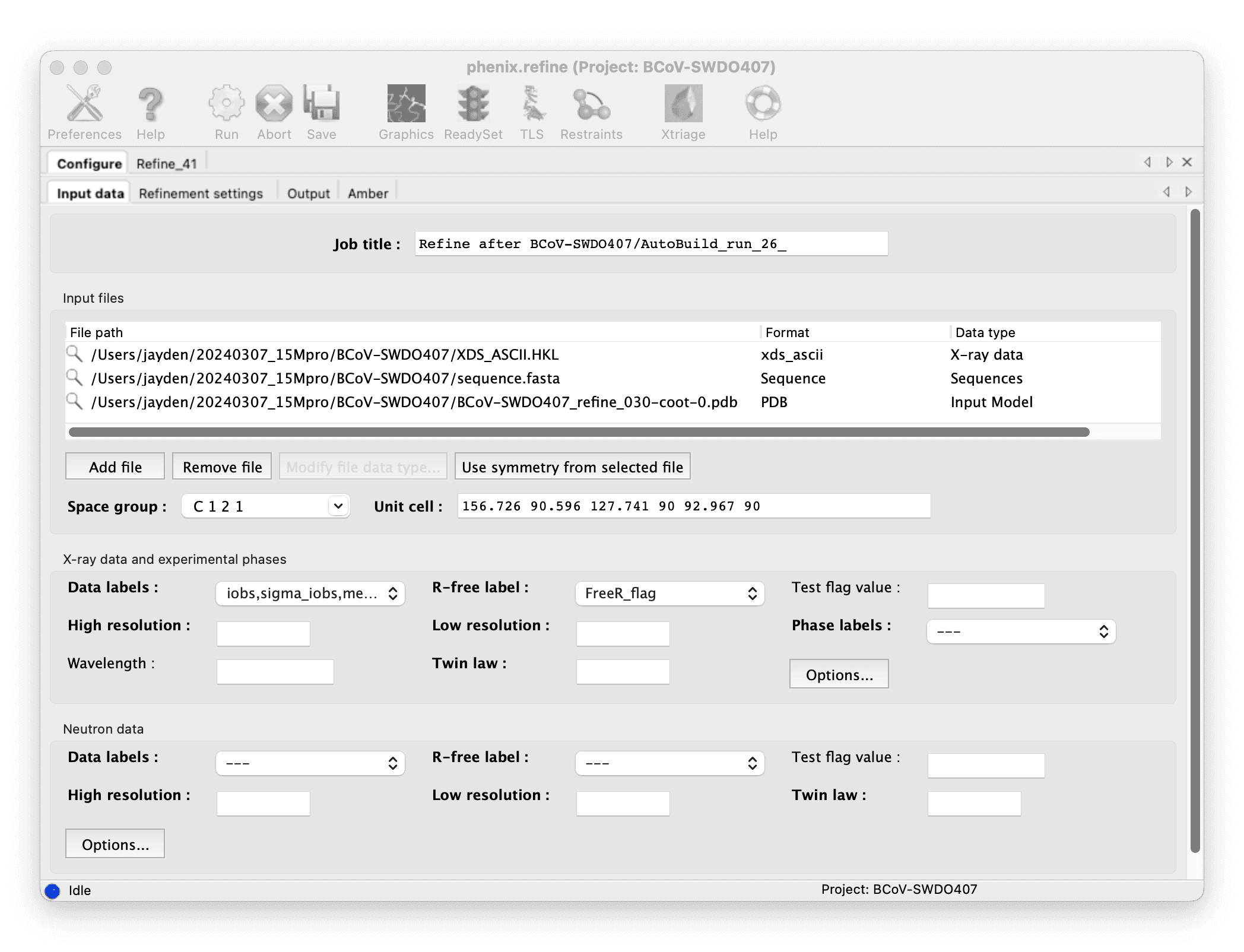
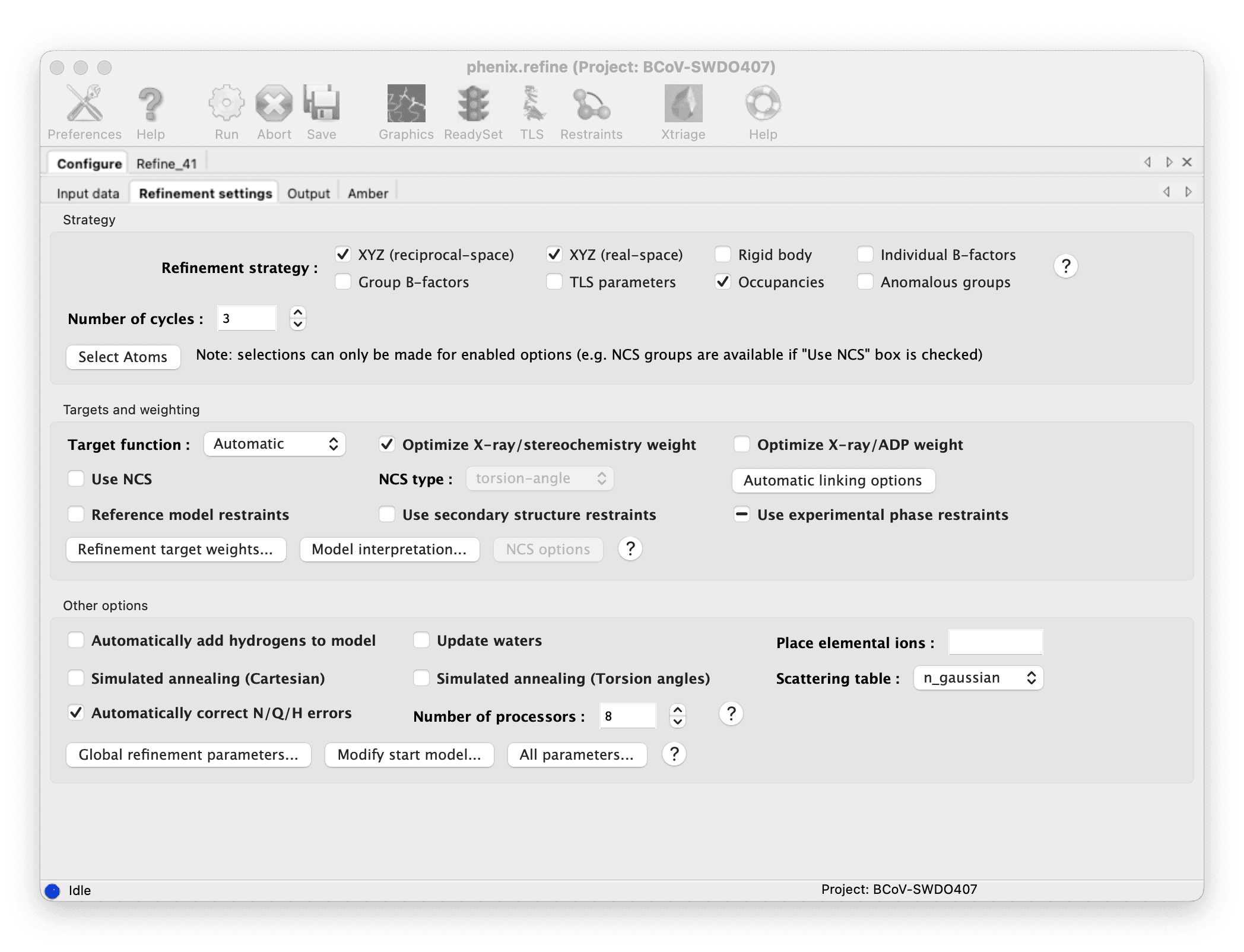
在 Coot 中检查 AutoBuild 的模型并手动精炼。
::: grid {cols=2,rows=1,gap=12,type=images}
:::
此文由 Mix Space 同步更新至 xLog
原始链接为 https://xxu.do/posts/academic/X-ray-data-processing